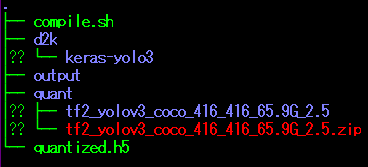第1部では、AIモデルのKeras変換、量子化、コンパイルを行うための環境構築や事前準備を行いました。第2部ではAIモデルのダウンロード~量子化/Keras変換/量子化/コンパイルまでを行っていきます。
AIモデルのインストール
今回使用するモデルはdarknet版のyolov3です。
C言語で書かれたオープンソースのニューラルネットフレームワークでインストールが簡単で試しやすいモデルとなっています。またネット上にも情報がたくさんあるため、今回はこのモデルを使用します。
「work/d2k/keras-yolov3」フォルダに以下コマンドでAIモデルをインストールします。
$ wget https://pjreddie.com/media/files/yolov3.weights
Keras変換
ダウンロードしてきたDarknetモデルをKeras形式に変換します。
-
①
-
変換する前にソースコードを修正します。
第1部コラムでダウンロードしたKeras変換ツールのソースコードconvert.pyではエラーが発生したため、以下に記載されているコードに置き換えます。
※補足)ダウンロードしたkeras-yolov3ツールで使用しているKerasは古いバージョンのため、下記ページのブロガーがtensorFlow 2.0に組み込まれているKerasバージョンをベースにconvert.pyを修正しています。
https://waittim.github.io/2020/10/25/darknet2keras/
-
②
-
以下コマンドで変換を行います。
※「model_data」フォルダを「convert.py」と同じディレクトリに作成しておくこと
$ python convert.py -cfg_path yolov3.cfg -weights_path yolov3.weights -output_path model_data/yolov3.h5
以下のようなログが出力されます
作成した「model_data」フォルダの中にファイルが作成されていたら変換成功です。
次は変換したモデルで量子化を行っていきます。
作業中に発生したValueErrorについて
Keras変換中に下記のようなValueErrorが発生した場合、
インストールしたライブラリ間で齟齬があることが原因なので、以下のコマンドを実施してライブラリのバージョンを変更します。
$ pip install tensorflow-gpu==2.8.0
量子化
Keras変換したモデルを量子化していきます。
-
①
-
量子化用データセット(coco2017)をダウンロード
Vitis AI での量子化は、ラベルのないデータセットが必要になるのでデータセットをダウンロードします。
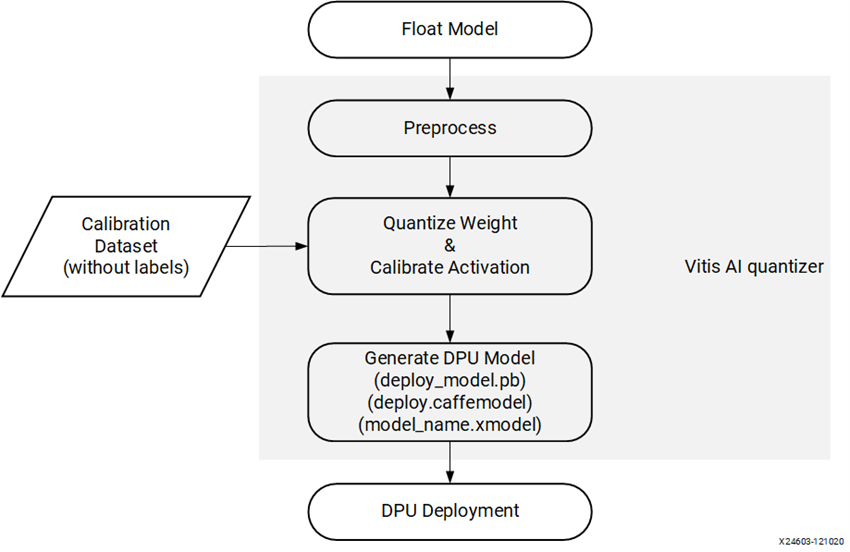
※以下は量子化のワークフロー図
1. download_data.shと同じディレクトリに「data」フォルダを作成します。
2. convert_data.shのスクリプト中のパスを変更します。(任意)
実際に使用したコード
3. 以下コマンドでダウンロードします。
$ bash download_data.sh
$ bash convert_data.sh
※download_data.sh、convert_data.shは構築した量子化環境にあります。
「data」フォルダにデータセットがダウンロードされると以下のようになります。
-
②
-
量子化用スクリプト(Run_eval.sh)中のパスやコマンドを修正します。
実際に変更した箇所は以下です。
・17行目
(変更前) WEIGHTS=../../float/yolov3.h5
(変更後) WEIGHTS=float/yolov3.h5
・18行目
(変更前) ANNO_JSON=../../data/annotations/instances_val2017.json
(変更後) ANNO_JSON=data/annotations/instances_val2017.json
・19行目
(変更前) ANNO_FILE=../../data/val2017.txt
(変更後) ANNO_FILE=data/val2017.txt
・25行目
(変更前) python eval.py … --save_result
(変更後) python eval.py … --save_result –quant
実際に使用したコード
ここからVitis Aiを使用して実際に量子化していきます。
-
③
-
dockerコンテナを起動
1. docker_run.sh を実行するためにVitis AIのディレクトリに移動します。
$ cd Vitis-AI
2. そして以下コマンドで起動します。
$ sudo ./docker_run.sh xilinx/vitis-ai-cpu:latest
以下のような出力で起動完了です。
3. 量子化環境を以下のコマンドで有効化します。
-
④
-
run_eval.shを実行するためカレントディレクトリを移動します。
$ cd model_zoo/work/quant/tf2_yolov3_coco_416_416_65.9G_2.5/code/test
-
⑤
-
floatフォルダにKeras変換手順で作成したyolov3.h5をコピーします。
-
⑥
-
以下コマンドで量子化実行します。
$ bash ./run_eval.sh
GPUがない場合、実行するとエラーがいくつか発生しますが無視で良いです。
※足りないライブラリは都度インストール
(実際に実施したコマンドは以下)
$ pip install MNN
$ pip install pycocotools
$ pip install onnxruntime
$ pip install imgaug
$ pip install keras_applications
$ pip install bokeh
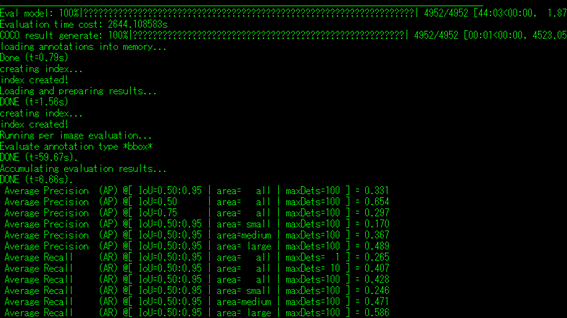
以下のようなログが出力されたら完了です。
「quantized」フォルダに量子化モデルが生成されていることを確認します。
次は量子化モデルをコンパイルしていきます。
〇作業中に発生したFileNotFoundErrorについて
量子化中に下記のFileNotFoundErrorが発生した場合、
「val2017.txt」に記載されているデータセットへのパスが正しくないことが原因です。
quant\tf2_yolov3_coco_416_416_65.9G_2.5\code\test\data\val2017.txt
量子化環境の有効状態と無効状態ではパスが異なることが分かりました。
量子化環境を有効にした状態でのパスに置換してください。
※cocoデータセットをインストールするときは量子化環境を有効にしてから実施するとエラーが発生しなかったかもしれません。
コンパイル
量子化したモデルをターゲットボード向けにコンパイルしていきます。
-
①
-
まずはコンパイルの実行ファイルを作成します。
$ touch comile.sh
今回は以下のディレクトリに作成します。
Vitis-AI/model_zoo/work/
-
②
-
作成したファイルに以下ソースコードをコピーし保存します。
$ vim compile.sh
※:wqで保存してvimを閉じます
以下のような構成でコンパイルしていきます。
※ここでは実行ファイルと同じディレクトリに量子化モデルをコピーしています
-
③
-
量子化環境を立ち上げます。
手順は量子化手順③と同じなので省略
-
④
-
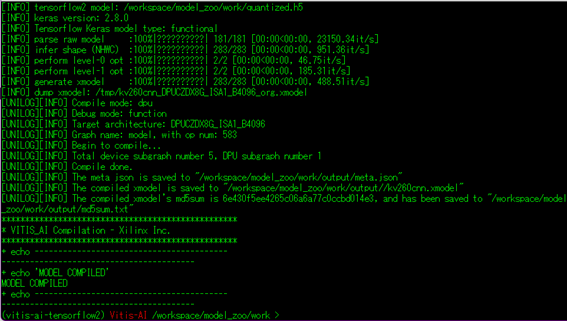
以下のコマンドでコンパイルを実行します。
$ bash -x compile.sh
完了すると以下のようなログが出力されます。
-
⑤
-
「output」フォルダにファイルが生成されていることを確認します。
無事にDPUで動かす「.xmodel」ファイルを生成することができました!
第3部では実際にDPUを使って推論を行い、比較検証を行います!
メーカー
AMD社の製品はこちら
関連ソリューション
FPGA/基板の受託開発
個別相談も承っております。下記よりお申し込みください。